- 카테고리 Genomics > Variant-analysis
- 수정일2025-10-30 10:46:04
- 레퍼런스
Bio-Express Somatic WGS Pipeline은 전장 유전체 시퀀싱 데이터로부터 체세포 변이를 검출하기 위한 모듈식 분석 파이프라인입니다. 이 파이프라인은 raw FASTQ 파일을 입력 데이터로 사용하고, 종양-정상 쌍 분석을 기반으로 하는 포괄적인 체세포 변이 호출 결과와 품질 평가 및 시각화를 제공합니다. FastQC를 통한 시퀀싱 품질 평가 후, Cutadapt로 어댑터 제거 및 품질 트리밍을 수행하고, BWA-MEM2 정렬 도구를 사용하여 참조 유전체 서열에 매핑하여 BAM 형식의 정렬 파일을 생성합니다. 이후 GATK 파이프라인을 통해 중복 제거, 매핑 품질 평가, 그리고 저품질 read에 대한 필터링을 수행하며 모든 페어 정보가 일치하는지 확인합니다. SAMtools를 활용한 좌표 기준 정렬과 GATK MarkDuplicates를 통한 PCR 중복 제거를 거쳐, GATK BaseRecalibrator와 ApplyBQSR을 사용하여 알려진 변이 사이트 정보를 공변량으로 활용한 염기 품질 점수 재보정을 수행합니다. 재보정이 완료된 BAM 파일에 대해 먼저 포괄적인 품질 관리 및 샘플 검증 단계를 수행합니다. Somalier를 통한 샘플 관계 검증, SNPmatch를 활용한 변이-SNP마커 통합 분석을 통한 샘플 정체성 확인, VerifyBamID2를 통한 샘플 오염도 평가, 그리고 Mosdepth를 사용한 커버리지 분석을 통해 시퀀싱 데이터의 품질과 신뢰성을 종합적으로 평가합니다. 이어서 종양-정상 쌍 분석 단계로 진입하며, Conpair를 통한 Normal-Tumor 페어 적합성 검증과 교차 개체 오염 수준 추정을 수행합니다. 그 다음 Strelka2와 Mutect2를 통한 단일 염기 변이 및 삽입/결손 변이 검출을 병행하여 체세포 변이의 민감도와 특이도를 극대화합니다. 마지막으로 TINC를 통한 종양 순도 분석과 Manta를 사용한 구조 변이 호출, Canvas를 이용한 복제수 변이 분석으로 포괄적인 체세포 유전체 변화를 정량화하여 암 유전체학 연구와 정밀 의학에 필수적인 정보를 제공합니다.
> 기본 참조 게놈: hg38
[중요] 샘플 유형 식별 방법:
- 종양 조직 샘플: FASTQ 파일명에 "_T" 포함 필수
- 정상 조직 샘플: FASTQ 파일명에 "_N" 포함 필수
(예시)
patient001_T_R1.fastq.gz # 종양 샘플, Read 1
patient001_T_R2.fastq.gz # 종양 샘플, Read 2
patient001_N_R1.fastq.gz # 정상 샘플, Read 1
patient001_N_R2.fastq.gz # 정상 샘플, Read 2
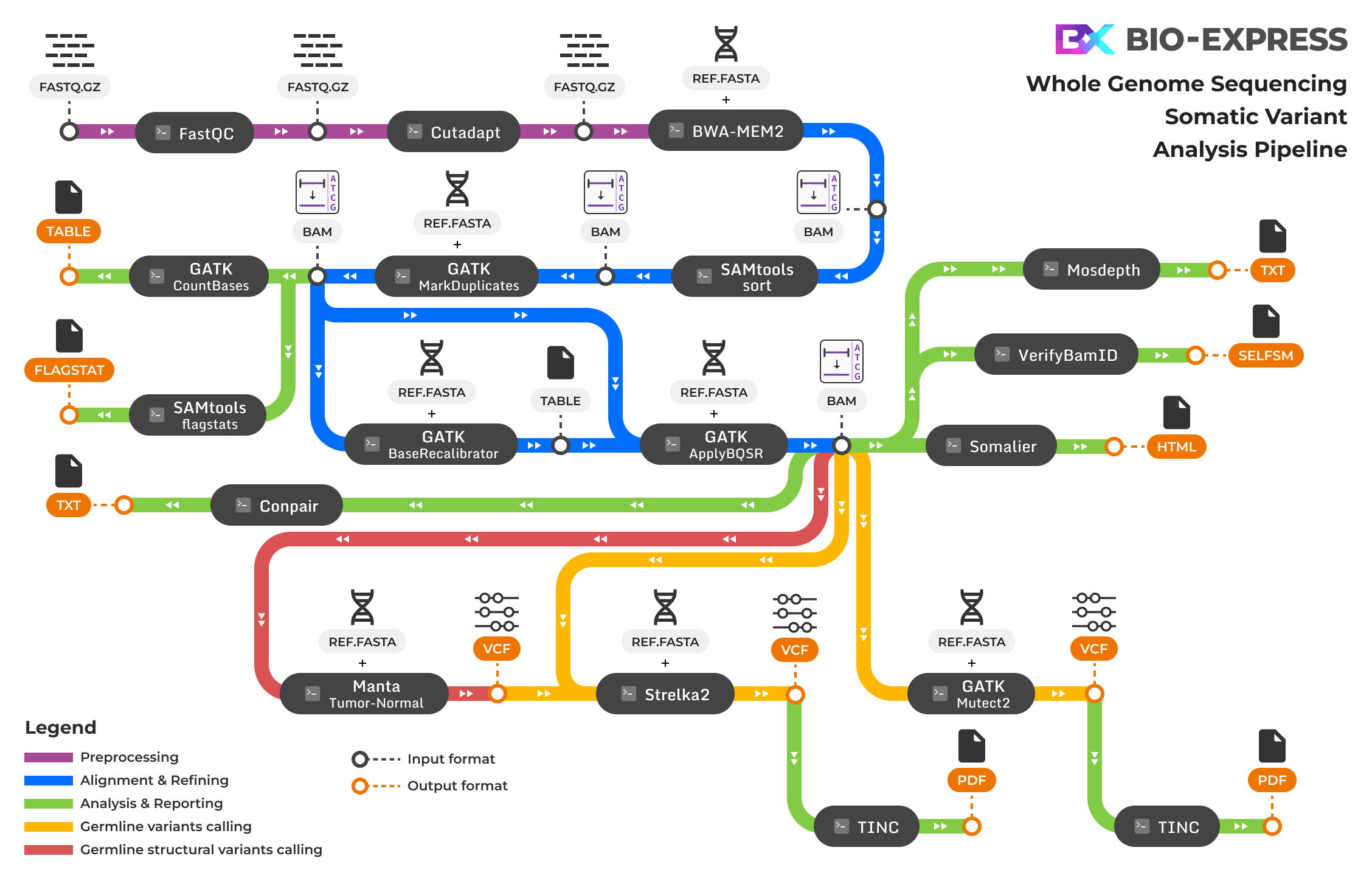
파이프라인 모듈
FastQC
FastQC는 고속 염기서열 분석(high throughput sequence) 데이터의 품질 관리를 위한 분석도구입니다. 이 프로그램은 FASTQ 형식의 서열 데이터를 읽어들여 여러 품질 관리(Qaulity Control) 검사를 수행하고 결과는 HTML 기반의 보고서로 출력합니다. FastQC는 전반적인 품질 문제에 대한 개요 정보를 제공하며, 쉽게 확인할 수 있는 요약된 그래프와 테이블을 포함합니다. FastQC는 FASTQ 형식의 파일이 입력 파일로 사용되며, 출력 결과는 리포트 html 파일과 zip 형식의 압축 파일이 생성됩니다.
주요사항
- FastQC는 자바 애플리케이션입니다. 실행하기 위해서는 시스템에 적절한 자바 실행 환경(Java Runtime Environment, JRE)이 설치되어 있어야 합니다. 따라서 FastQC를 실행하기 전에 먼저 적절한 JRE가 설치되어 있는지 확인해야 합니다. 다양한 종류의 JRE를 사용할 수 있지만, 저희가 테스트해본 것은 최신 오라클 런타임 환경과 adoptOpenJDK 프로젝트의 JRE입니다. 64비트 JRE를 다운로드하여 설치하고, 자바 애플리케이션이 시스템 경로(path)에 포함되도록 설정해야 합니다(대부분의 설치 프로그램이 이를 자동으로 처리해줍니다).
실행 명령어 예시
$program_dir/fastqc –t 6 –o $OUTPUT_DIR $INPUT_DIR/$READ
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | FASTQ 형식의 파일이 있는 디렉토리 경로 | |
| Output | Folder | output_dir | path/to/output_dir | 실행 후 결과물을 저장할 디렉토리 경로 (-o) |
결과
Cutadapt
Cutadapt는 NGS 시퀀싱 데이터에서 어댑터(adapter) 서열 제거, 품질이 낮은 서열 제거 등 다양한 전처리 작업을 수행하는 데 사용되는 도구입니다. 주로 서열의 3’ 말단에서 염기서열을 제거하며, 특히 RNA 시퀀싱 또는 DNA 시퀀싱 데이터에서 어댑터 제거에 빈번하게 활용됩니다. 전체 데이터의 품질을 유지하여 이후의 분석 단계에서 정확한 결과를 얻을 수 있도록 도움을 줍니다. 입력 데이터로는 fastq, fastq.gz, fq, fq.gz 확장자를 가진 파일을 사용할 수 있습니다. 출력으로는 파일 확장자 앞쪽에 ‘_trimmed’라는 문구가 붙은 fastq 파일이 생성되도록 세팅되어 있습니다. 따라서 "trimmed.fastq" 파일은 프라이머 잔여물이나 다른 불순물이 제거된 reads 파일을 나타냅니다.
주요사항
- 1. Bio-Express의 Cutadapt 모듈은 paired-end와 single-end 형식의 FASTQ 데이터를 모두 처리할 수 있도록 설계되었습니다.
- 2. -a 및 –A 옵션에 입력해야 할 각각의 어댑터 서열은 NGS 시퀀서 플랫폼별로 다음과 같습니다. > Illumina: 기본값 -a AGATCGGAAGAGCACACGTCTGAACTCCAGTCAC -A AGATCGGAAGAGCGTCGTGTAGGGAAAGAGTGT > MGI: 필요시 기본값 대체 입력 -a AAGTCGGAGGCCAAGCGGTCTTAGGAAGACAA -A AAGTCGGATCGTAGCCATGTCGTTCTGTGAGCCAAGGAGTTG
실행 명령어 예시
$PROGRAM_DIR/cutadapt -q 20 –Q 20 -j 6 -a AGATCGGAAGAGCACACGTCTGAACTCCAGTCAC -A AGATCGGAAGAGCGTCGTGTAGGGAAAGAGTGT --minimum-length $MIN_LEN_R1:$MIN_LEN_R2 --pair-filter=$PAIR_FILTER -o $OUTPUT_DIR/$READ_1 –p $OUTPUT_DIR/$READ_2 $INPUT_DIR/$READ_1 $INPUT_DIR/$READ_2
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | FASTQ 형식의 파일이 있는 디렉토리 경로 | |
| Output | Folder | output_dir | path/to/output_dir | 실행 후 결과물을 저장할 디렉토리 경로 (-o, -p) | |
| Option | Integer | min_len_r1 | 70 | --minimum-length $MIN_LEN_R1:$MIN_LEN_R2 어댑터 제거 후 남아 있는 Read1의 최소 길이 | |
| Option | Integer | min_len_r2 | 1 | --minimum-length $MIN_LEN_R1:$MIN_LEN_R2 어댑터 제거 후 남아 있는 Read2의 최소 길이 | |
| Option | String | pair_filter | any | --pair-filter (single-end FASTQ에는 미적용) - any: 두 Read 중 어느 하나라도 조건에 부합하면 필터링 - both: 두 Read 모두 조건에 부합하면 필터링 | |
| Option | String | adapter_pos | adapter | 어댑터 시퀀스 처리 위치 지정 --adapter: 서열의 3' 끝 방향에서 어댑터를 찾아 해당 어댑터와 그 이후 모든 서열을 제거 (-a, -A) --front: 서열의 5' 시작 방향에서 어댑터를 찾아 해당 어댑터와 그 이전 모든 서열을 제거 (-g, -G) --anywhere: 5' 또는 3' 어느 쪽에든 나타날 수 있는 어댑터를 탐지 (-b, -B) | |
| Option | String | adapter_r1 | AGATCGGAAGAGCACACGTCTGAACTCCAGTCAC | 첫 번째 Read의 어댑터 시퀀스 (-a) | |
| Option | String | adapter_r2 | AGATCGGAAGAGCGTCGTGTAGGGAAAGAGTGT | 두 번째 Read의 어댑터 시퀀스 (-A) | |
| Option | Integer | quality_r1 | 20 | 첫 번째 Read의 절단 기준으로 사용할 품질 임계값 (-q) | |
| Option | Integer | quality_r2 | 20 | 두 번째 Read의 절단 기준으로 사용할 품질 임계값 (-Q) |
결과
BWA-MEM
BWA는 대용량 시퀀싱 데이터를 참조 유전체와 정렬하는 데 사용되는 고속, 고정밀 분석 도구입니다. BWA는 짧은 리드(30bp~1Mbp)와 긴 리드(>100bp)에 모두 적합하며, 주로 Illumina 시퀀싱 데이터를 정렬하는 데 사용됩니다. BWA의 주요 기능은 효율적인 시퀀스 정렬도, Burrows-Wheeler 변환과 FM 인덱스를 사용하여 시퀀싱 리드를 참조 유전체에 빠르고 정확하게 정렬합니다. BWA-bactrack, BWA-sw 및 BWA-mem의 세 가지 알고리즘으로 구성됩니다. BWA-mem은 긴 리드(>70bp)에 최적화되어 있습니다. 시드-확장 접근 방식을 사용하여 정확하고 효율적인 정렬을 수행하며, 특히 긴 리드에서 높은 성능을 보입니다. 또한 BWA-mem은 RNS-seq 데이터에서 인트론과 엑손 간의 정확한 정렬을 지원하며, 높은 민감도와 정확도를 가지고 있어 변이 탐지에 유리합니다. Bio-Express의 Whole Genome Sequencing Pipeline 분석과정에서 BWA-mem의 입력(input) 데이터로는 fastq 타입의 파일이 가능하며, 출력 데이터로는 align이 완료된 sam 파일이 생성됩니다.
주요사항
- 1. '-R <문자열>'에서는 완전한 Read Group 헤더 라인을 지정해야 합니다. 이는 후속 분석에서 샘플을 구별하는 데 매우 중요하며, Bio-Express 모듈에서는 아래와 같은 규칙으로 자동 입력됩니다. -R "@RG\\tID:${clean_name}\\tSM:${clean_name}\\tPL:ILLUMINA\\tLB:UNKNOWN" ${clean_name}: '_trimmed', '_R1', '_R2' 등과 같은 불필요한 접미사들은 제거된 샘플명 2. Reference FASTA 파일의 BWA-MEM 프로그램 전용 index가 존재하지 않는다면, reference FASTA 파일로부터 해당 index를 자동으로 생성하여 진행시키도록 설계되어 있습니다. 3. 입력 파일로 사용되는 FASTQ 파일명에 '_trimmed' 또는 '_HQ'와 같은 문구가 있을 경우, 결과 파일명에서 해당 문구는 제거됩니다.
실행 명령어 예시
#BWA-MEM $PROGRAM_DIR/bwa mem –M -v 3 –t 6 –K 100000000 –R "@RG\\tID:${clean_name}\\tSM:${clean_name}\\tPL:ILLUMINA\tLB:UNKNOWN" $REFERENCE_GENOME $READ_1 $READ_2 > $OUTPUT_SAM
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | /path/to/input_dir | FASTQ 형식의 파일이 있는 디렉토리 경로 | |
| Output | Folder | output_dir | /path/to/output_dir | Reference genome FASTA에 FASTQ 데이터가 매핑되어 생성된 SAM/BAM 파일이 생성될 경로 | |
| Option | File | ref_genome | hg38 | Reference genome FASTA 파일. BWA index의 basename. |
결과
SAMtools_sort
SAMtools는 고처리량 시퀀싱 데이터를 조작하고 분석하기 위해 설계된 다목적 도구 모음으로, 특히 SAM, BAM, CRAM 형식의 정렬 데이터를 처리하는 데 사용됩니다. 이 도구는 변이 호출, 정렬 데이터 확인, 품질 관리 등 다양한 작업에 널리 활용됩니다. SAMtools는 인덱싱, 정렬, 병합, 필터링 등 정렬 파일 처리에 필요한 다양한 기능을 지원하며, 유전체 분석 파이프라인에서 필수적인 도구입니다.SAMtools sort 명령은 SAM, BAM, CRAM 파일을 지정된 기준에 따라 정렬합니다. 기본적으로 좌표(coordinate) 기준으로 정렬하며, -n 옵션을 사용하면 쿼리 이름(query name) 기준으로 정렬합니다. 정렬 순서는 SAM 파일 헤더의 @HD 태그 내 SO 필드에 표시됩니다. 좌표 정렬은 @HD SO:coordinate, 쿼리 이름 정렬은 @HD SO:queryname으로 헤더에 기록됩니다.
좌표 정렬 (Coordinate Sorting)의 경우, 읽기는 다음과 같은 순서로 정렬됩니다:
1. 참조 시퀀스 이름(RNAME): @SQ 태그에 정의된 참조 시퀀스 사전의 순서를 따릅니다.
2. 가장 왼쪽 매핑 위치(POS): 동일한 RNAME 내에서 읽기의 시작 위치를 기준으로 정렬됩니다.
3. REVERSE 플래그: POS가 동일한 경우, 순방향 가닥(forward strand, REVERSE 플래그 0)이 역방향 가닥(reverse strand, REVERSE 플래그 1)보다 먼저 옵니다.
이후 추가적인 동점이 있는 경우, 정렬 순서는 입력 데이터의 순서를 유지할 수 있습니다.
쿼리 이름 정렬 (Query Name Sorting)의 경우, -n 옵션을 사용하며 읽기는 다음과 같은 순서로 정렬됩니다:
1. 쿼리 이름(QNAME): 자연스러운 순서(natural order)로 정렬되며, 문자열 내 숫자 부분은 수치적으로 비교됩니다 (예: "read9"는 "read10"보다 먼저).
2. READ1/READ2 플래그: 동일한 QNAME을 가진 읽기는 첫 번째 읽기(READ1, 플래그 0x40)가 두 번째 읽기(READ2, 플래그 0x80)보다 먼저 옵니다.
3. 정렬 유형: READ1/READ2가 동일한 경우, 주 정렬(primary alignment)이 먼저 오고, 그 다음 보조 정렬(supplementary alignment), 2차 정렬(secondary alignment) 순으로 정렬됩니다.
남은 동점은 입력 데이터의 원래 순서를 따릅니다. 따라서 SAMtools는 유전체 데이터를 효율적으로 처리하고 분석하는 데 중요한 역할을 하며, 특히 정렬 작업에서 sort 도구가 유용하게 사용됩니다. Bio-Express의 Whole Genome Sequencing Pipeline 분석과정에서 SAMtools sort는 SAM파일을 입력 데이터로 사용하여 BAM 파일을 출력합니다.
실행 명령어 예시
$PROGRAM_DIR/samtools sort -@ 6 –O bam —write-index –o $OUTPUT_BAM $INPUT_BAM
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | /path/to/input_dir | Sorting이 필요한 BAM 파일을 찾을 디렉토리 | |
| Output | Folder | output_dir | /path/to/output_dir | Sorting이 완료된 BAM 파일을 저장할 디렉토리 |
결과
GATK MarkDuplicates
GATK의 MarkDuplicates는 BAM 또는 SAM 파일에서 중복 리드를 식별하고 태그하는 분석 도구입니다. BAM/SAM 파일에서 리드와 리드 페어의 5' 위치의 시퀀스를 비교하여 중복을 식별합니다. 중복된 리드를 모은 후에는 리드의 베이스 품질 점수의 합을 기준으로 주 리드와 중복 리드를 구별하는 알고리즘을 사용하여 표시합니다. 이러한 중복 서열은 변이 검출 시 위양성(false positive)을 유발할 수 있으므로, 제거하거나 통계 분석에서 제외하는 것이 중요합니다. Bio-Express의 Whole Genome Sequencing Pipeline 분석과정에서 GATK의 MarkDuplicates의 입력(input) 데이터로는 BAM 파일이며, 중복이 식별된 리드가 표시된 새로운 BAM 파일과 중복된 리드의 수와 관련된 다양한 통계를 담은 메트릭스 파일을 출력(output) 데이터로 합니다.
실행 명령어 예시
$PROGRAM_DIR/gatk MarkDuplicates \ -I $INPUT_BAM -O $OUPUT_BAM -R $REFERENCE_GENOME -M $METRICS_file
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | 중복 서열 제거 처리 대상 BAM 파일을 찾을 디렉토리 | |
| Output | Folder | output_dir | path/to/output_dir | Sorting이 완료된 BAM 파일을 저장할 디렉토리 | |
| Option | File | ref_genome | hg38 | Reference genome FASTA 파일 |
결과
GATK CountBases
GATK의 CountBases는 BAM 또는 SAM 파일에서 각 염기(A, C, G, T)와 기타 염기 외 문자(N 등)의 출현 빈도를 계수하는 분석 도구입니다. 시퀀싱 데이터의 품질을 평가하거나 유전자 발현 수준, 변이 분석 등 다양한 분석 과정에서 유용하게 사용됩니다. CountBases의 출력은 초기 데이터 탐색 단계에서 데이터의 일관성과 정확성을 평가하는 데 중요한 지표를 제공하며, 이를 기반으로 추가적인 실험 설계나 분석 전략을 결정할 수 있습니다. Bio-Express의 Whole Genome Sequencing Pipeline 분석과정에서 GATK의 CountBases는 입력(input) 데이터로 BAM 파일을, 출력(output) 데이터로는 BAM 파일 전체 bases 수가 출력된 count 파일이 생성됩니다.
실행 명령어 예시
$program_dir/gatk CountBases -I $INPUT_BAM -O $OUTPUT_DIR -R $REFERENCE_GENOME -L $GENOMIC_INTERVALS -RF NotSecondaryAlignmentReadFilter
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | 염기 개수를 계산할 BAM 파일을 찾을 디렉토리 | |
| Output | Folder | output_dir | path/to/output_dir | 염기 개수 계산 결과물을 저장할 디렉토리 | |
| Option | File | ref_genome | hg38 | Reference genome FASTA 파일 (-R) | |
| Option | File | genomic_intervals | hg38 | Genomic Interval 파일: 본 분석 과정을 수행할 특정 유전체 영역을 지정 (-L) |
결과
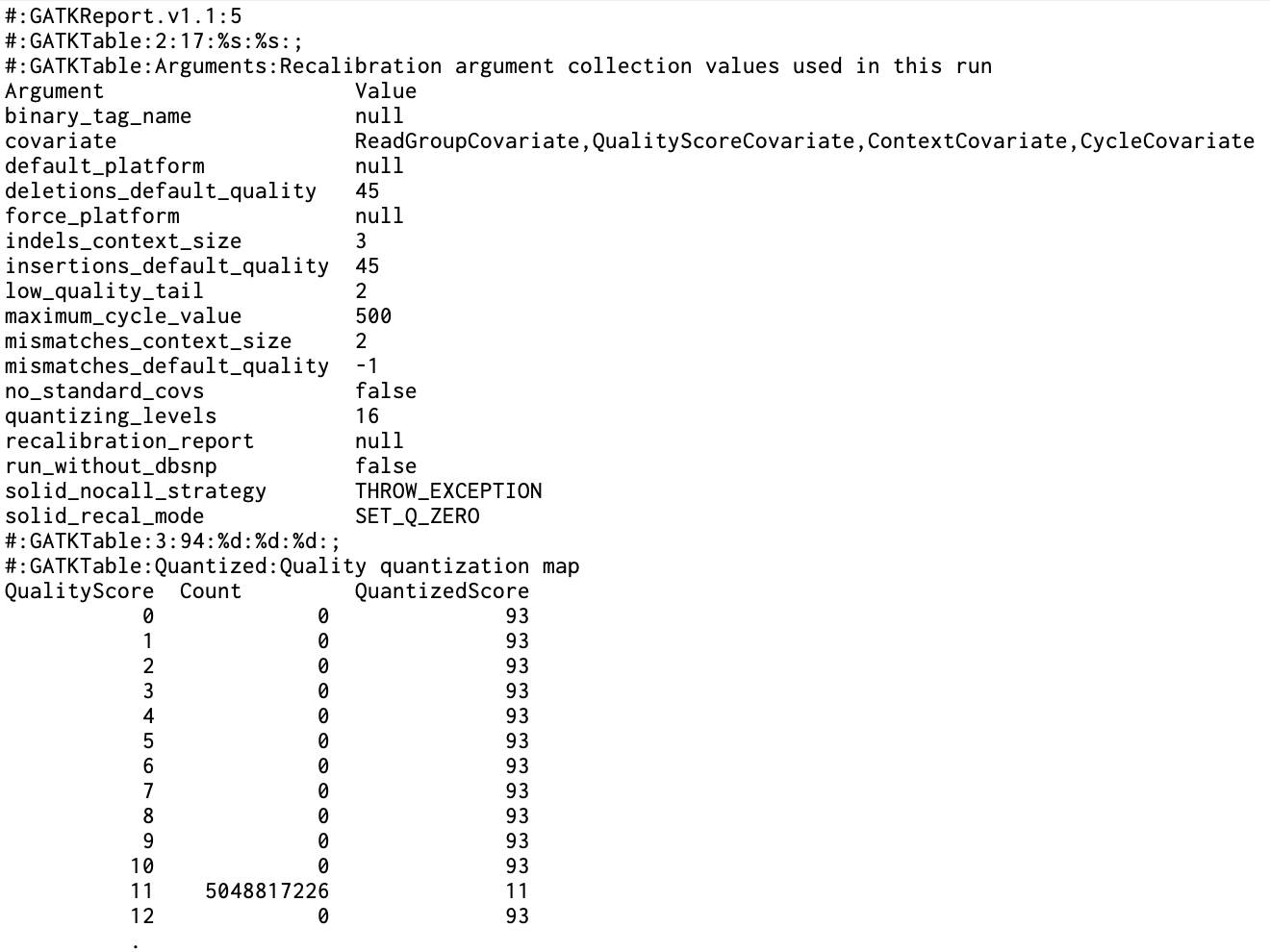
GATK BaseRecalibrator
GATK의 BaseRecalibrator는 리드의 염기 품질 점수와 오류 패턴을 모델링하기 위한 데이터를 수집합니다. 이 단계에서는 BAM 파일과 신뢰할 수 있는 변이 목록을 입력으로 받아, 리드의 품질 점수를 재조정하는 데 필요한 정보를 수집합니다. BaseRecalibrator는 다양한 공변랑을 기반으로 테이블을 생성합니다. 탐색은 알려진 변이 사이트 VCF 파일에 있는 위치에만 작동합니다. 모든 참조 불일치를 오류로 간주하고, 이를 통해 낮은 베이스 품질을 나타낸다고 가정합니다. 충분한 데이터를 통해 특정 공변량이 관찰된 사이트에서 경험적 오류 확률을 계산할 수 있습니다. 오류 확률은 불일치 횟수를 관찰 횟수로 나누어 계산됩니다. Bio-Express의 Whole Genome Sequencing Pipeline 분석과정에서 GATK의 BaseRecalibrator는 입력(input) 데이터로 BAM 파일, 참조 유전체 서열을 포함하는 FASTA 파일과 신뢰할 수 있는 변이 정보를 포함하는 VCF 파일입니다. 출력(output) 데이터로는 여러 공변량 값, 관찰 횟수, 불일치 횟수 그리고 경험적 품질 점수를 포함하는 table 파일입니다.
주요사항
- > hg38 reference genome 기준, 일반적으로 사용되는 known sites 4개 --known-sites dbsnp_138.hg38.vcf.gz --known-sites Mills_and_1000G_gold_standard.indels.hg38.vcf.gz --known-sites 1000G_phase1.snps.high_confidence.hg38.vcf.gz --known-sites Homo_sapiens_assembly38.known_indels.vcf.gz
실행 명령어 예시
$PROGRAM_DIR/gatk BaseRecalibrator -I $INPUT_BAM -O $OUTPUT_TABLE -R $REFERENCE_GENOME --known-sites $KNOWN_SITES_[1234]
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | BAM 파일이 있는 디렉토리 경로 | |
| Output | Folder | output_dir | path/to/output_dir | 실행 후 결과물을 저장할 디렉토리 경로 | |
| Option | File | ref_genome | hg38 | Reference sequence FASTA 파일 경로 | |
| Option | File | known_sites_1~4 | hg38 | known polymorohic sites인 VCF 파일 경로 |
결과
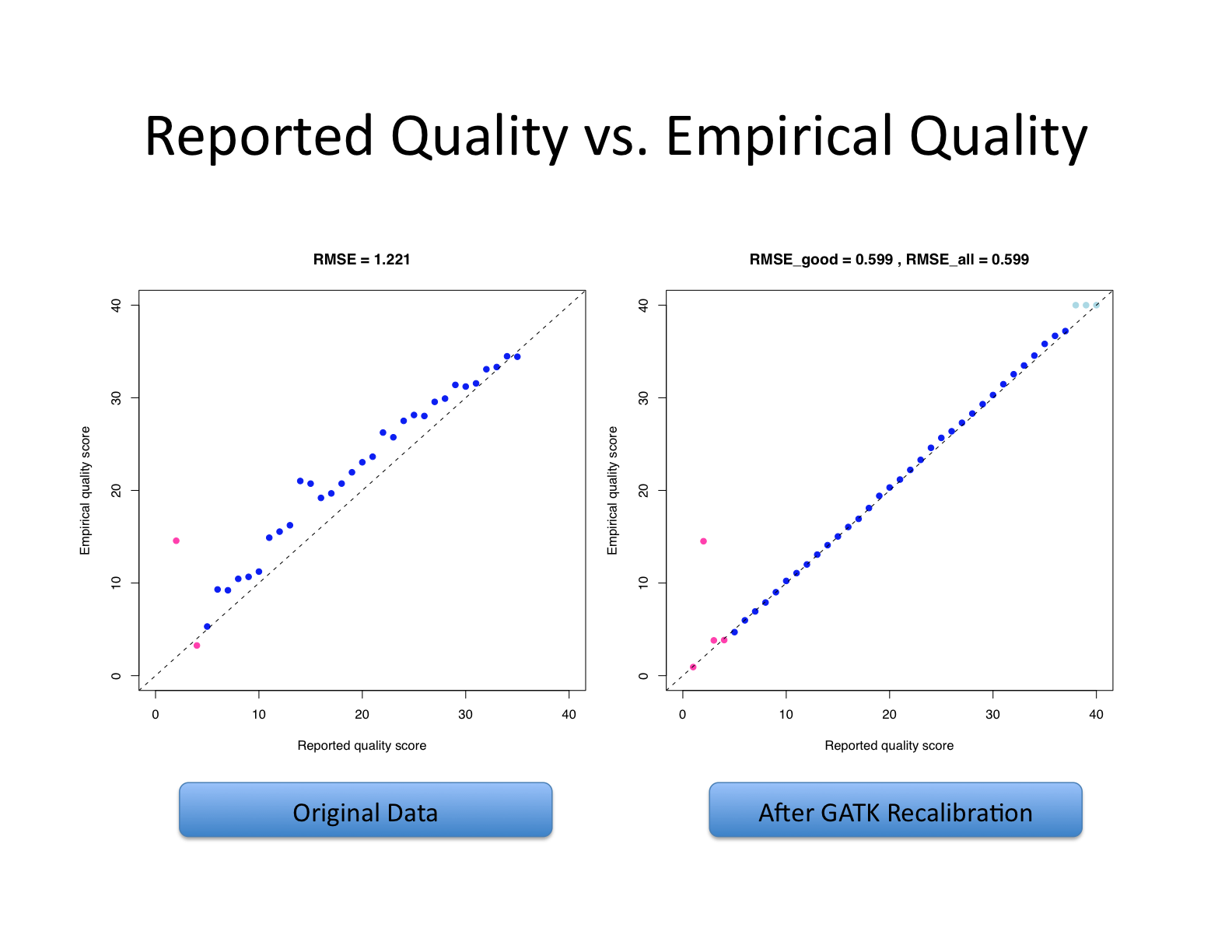
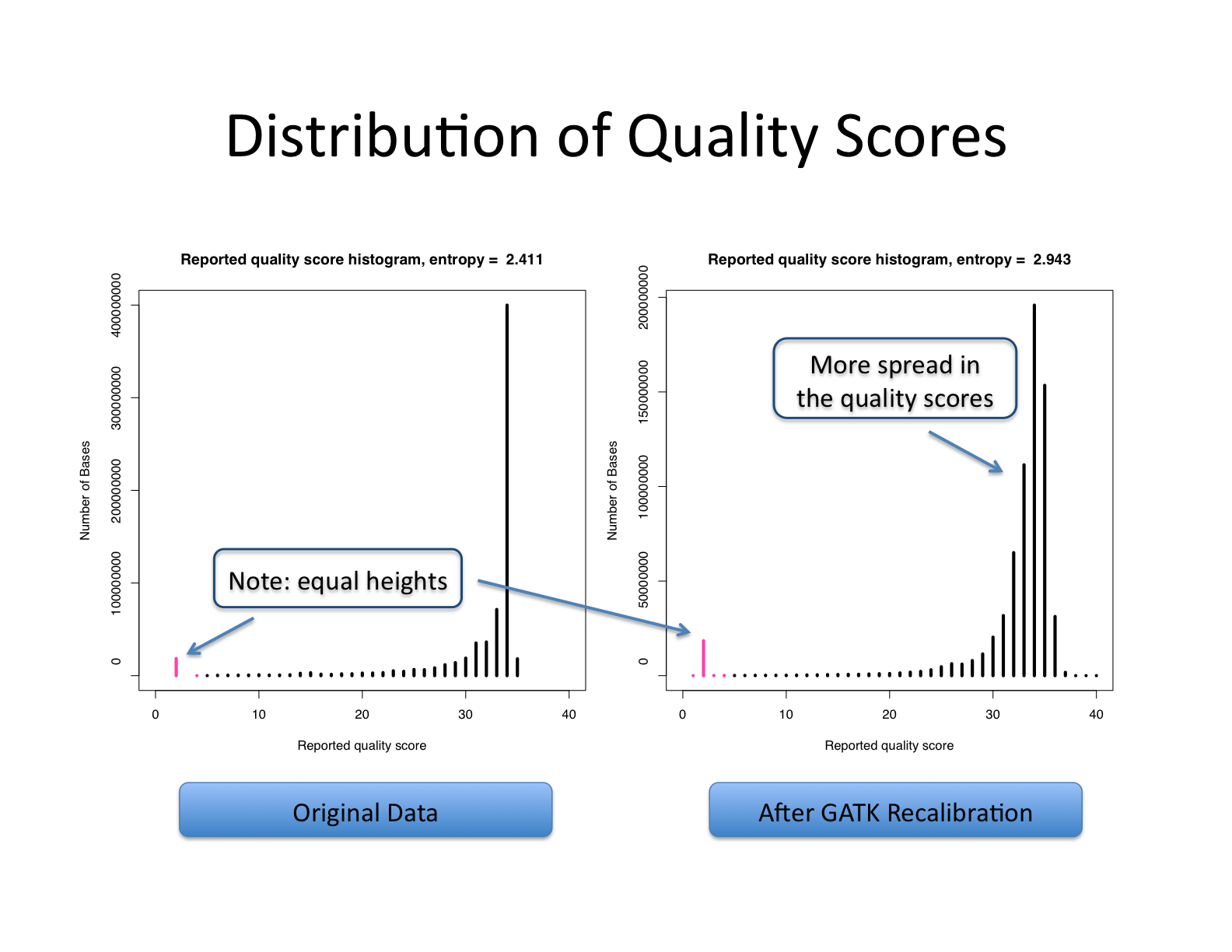
GATK ApplyBQSR
GATK의 ApplyBQSR은 GATK에서 중요한 역할을 하는 도구로, 시퀀싱 데이터의 염기 품질 점수를 재조정하여 변이 탐지의 정확성을 향상시키는 기능을 수행합니다. 이 도구는 “BaseRecalibrator”에서 생성된 재조정 테이블에 포함된 여러 공변량을 사용하여 BAM 파일의 각 염기에 대한 품질 점수를 정밀하게 재조정합니다. 기본 공변량에는 리드 그룹, 초기 품질 점수, 시퀀싱 사이클, 뉴클레오타이드 컨텍스트 등이 포함됩니다. 각 공변량을 통해 특정 염기에서 발생할 수 있는 오류 확률을 계산하고, 이를 바탕으로 새로운 품질 점수를 할당합니다. Bio-Express의 Whole Genome Sequencing Pipeline 분석과정에서 GATK의 ApplyBQSR은 입력(input) 데이터로 정렬된 BAM 파일, “BaseRecalibrator”를 통해 생성된 재조정 테이블 파일과 참조 유전체 서열을 포함하는 FASTA 파일을 사용합니다. 출력(output) 데이터로는 재조정된 품질 점수를 반영한 새로운 BAM 파일입니다.
실행 명령어 예시
$PROGRAM_DIR/gatk ApplyBQSR -I $INPUT_BAM -bqsr $RECAL_TABLE -O $OUTPUT_BAM -R $REFERENCE_GENOME
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | BAM 파일이 있는 디렉토리 경로 | |
| Input | Folder | table_dir | path/to/table_dir | BaseRecalibrator에 의해 table 파일이 생성된 경로 | |
| Output | Folder | output_dir | path/to/output_dir | 실행 후 결과물을 저장할 디렉토리 경로 | |
| Option | File | ref_genome | hg38 | Reference sequence FASTA 파일 경로 |
결과
Somalier
Somalier는 유전체 데이터의 샘플 간 관련성을 분석하고 확인하는 분석 도구입니다. 주로 샘플의 신원을 확인하고 유전체 데이터에서 샘플 간의 혼합이나 오류를 탐지하는 데 사용됩니다. BAM, CRAM, VCF 파일에서 정보 추출을 수행하며, 샘플 간의 유사도 매트릭스를 계산하여 시각화할 수 있습니다. Bio-Express의 Whole Genome Sequencing Pipeline 분석과정에서 Somalier는 입력(input) 데이터로 BAM 파일, Reference sequence FASTA 파일과 추출할 variant의 sites인 VCF 파일을 사용합니다. 출력(output) 데이터로는 추출된 샘플 정보가 포함된 JSON 형식의 .somalier 파일입니다.
주요사항
- > Relatedness의 의미 - 1.00 : 일란성 쌍둥이/클론/중복 샘플 - 0.50 : 부모-자식/형제자매/부모의 일란성 쌍둥이/일란성 쌍둥이의 자식 - 0.35 : 3/4 형제 (예: 같은 어머니, 아버지들이 형제) - 0.25 : 조부모-손자/이복형제/삼촌-조카/이종사촌 - <0.20 : 원거리 친척(사촌)/혈연 관계 없음
실행 명령어 예시
$program_dir/somalier extract –d $OUTPUT_DIR \ -s $SOMALIER_SITES \ -f $REFERENCE_GENOME \ $INPUT_BAM $program_dir/somalier relate –o $OUTPUT_DIR \ $SOMALIER_EXTRACT_OUTPUT
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | 분석할 BAM 파일이 있는 디렉토리 경로 | |
| Output | Folder | output_dir | path/to/output_dir | 실행 후 결과물을 저장할 디렉토리 경로 | |
| Option | File | ref_genome | hg38 | Reference sequence FASTA 파일 경로 | |
| Option | File | somalier_sites | hg38 | 추출할 variant의 sites인 VCF 파일 경로 |
결과
VerifyBamID
VerifyBamID2는 시퀀싱 데이터(BAM/CRAM 파일)에서 DNA 오염도(contamination)를 정확하게 추정하기 위한 차세대 품질 관리 도구입니다. 혈통에 무관한(ancestry-agnostic) 방법을 사용하여 다양한 인종 배경의 샘플에서도 일관되고 신뢰할 수 있는 오염도 측정을 제공합니다. 이 도구는 주성분 분석(Principal Component Analysis, PCA)과 특이값 분해(Singular Value Decomposition, SVD) 기법을 활용하여 의도된 샘플(intended sample)과 오염 소스(contaminating sample)의 유전적 특성을 각각 추정합니다. VerifyBamID2는 베이지안 최적화 알고리즘을 통해 두 소스의 주성분 좌표(PC coordinates)를 동시에 추정하고, 최대우도법(Maximum Likelihood Estimation)을 사용하여 오염 수준(Alpha)을 정량화합니다. VerifyBamID2의 핵심 혁신은 집단 구조(population structure)에 의존하지 않는 분석 방법입니다. 기존 도구들이 특정 인종 집단의 참조 패널에 의존했던 것과 달리, 이 도구는 SVD 기반의 차원 축소를 통해 샘플의 유전적 배경을 자동으로 추론하고 보정합니다. 이를 통해 혼혈 샘플이나 희귀 집단에서도 정확한 오염도 측정이 가능하며, 대규모 다인종 코호트 연구에서 특히 유용합니다. 특히 종양-정상 쌍 분석에서 VerifyBamID2는 정상 샘플에 종양 세포가 혼입되었는지, 또는 종양 샘플에 정상 세포가 과도하게 포함되었는지를 정량적으로 평가할 수 있습니다. 또한 샘플 라벨링 오류, 교차 오염(cross-contamination), 그리고 액체 생검(liquid biopsy)에서의 순환 종양 DNA(ctDNA) 비율 추정에도 활용됩니다.
주요사항
- > SVD_PREFIX 예시: - 1000g.phase3.100k.b38.vcf.gz.dat : hg38 기반 1000 Genomes - 1000g.phase3.100k.b37.vcf.gz.dat : hg19 기반 1000 Genomes - hgdp.100k.b37.vcf.gz.dat : hg19 기반 Human Genome Diversity Project SVD_PREFIX를 1000g.phase3.10k.b38.vcf.gz.dat으로 지정하고자 할 때, 1000g.phase3.10k.b38.vcf.gz.dat.V를 선택하면, “.V”가 자동으로 제거되도록 설계되어 있음.
- > 오염도 평가 기준 - FREEMIX < 0.02 : 정상 범위 - 0.02 ≤ FREEMIX < 0.05 : 경미한 오염 - FREEMIX ≥ 0.05 : 심각한 오염 (분석 주의)
실행 명령어 예시
$PROGRAM_DIR/verifybamid --SVDPrefix $SVD_PREFIX \ --BamFile $INPUT_BAM \ --Reference $REFERENCE_GENOME \ --Output $OUTPUT_PREFIX \ --NumThread 6
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | BAM 파일이 있는 디렉토리 경로 | |
| Output | Folder | output_dir | path/to/output_dir | 실행 후 결과물을 저장할 디렉토리 경로 | |
| Option | File | ref_genome | hg38 | Reference sequence FASTA 파일 경로 | |
| Option | File | svd_prefix | 1000g.phase3.10k.b38.vcf.gz.dat.V | Singular Value Decomposition(SVD) 기법으로 준비된, VerifyBamID 전용 참조 패널 파일들(.UD, .mu, .bed, .V)의 prefix |
결과
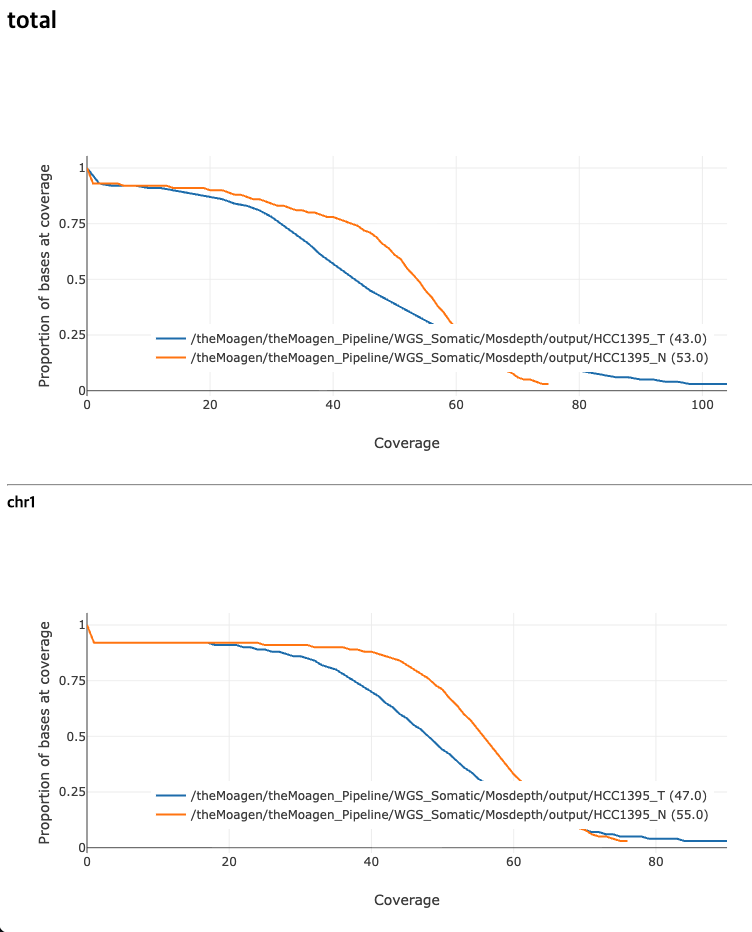
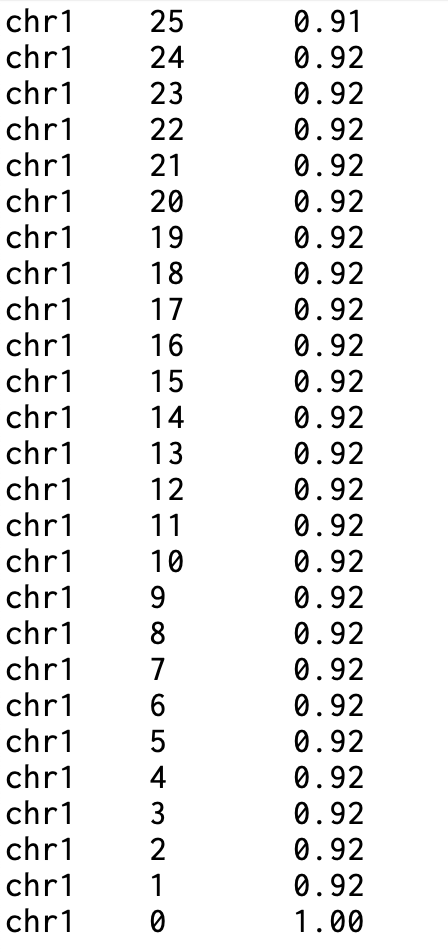
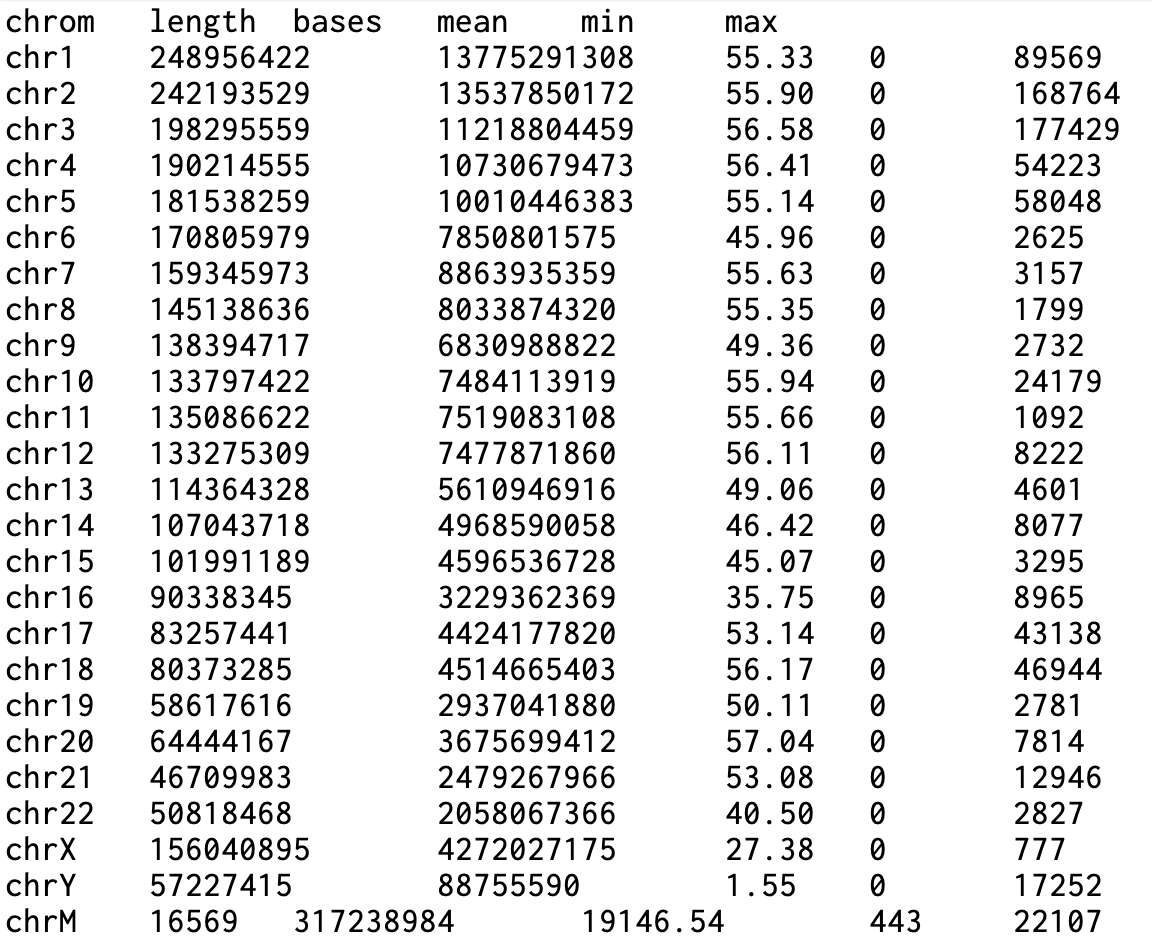
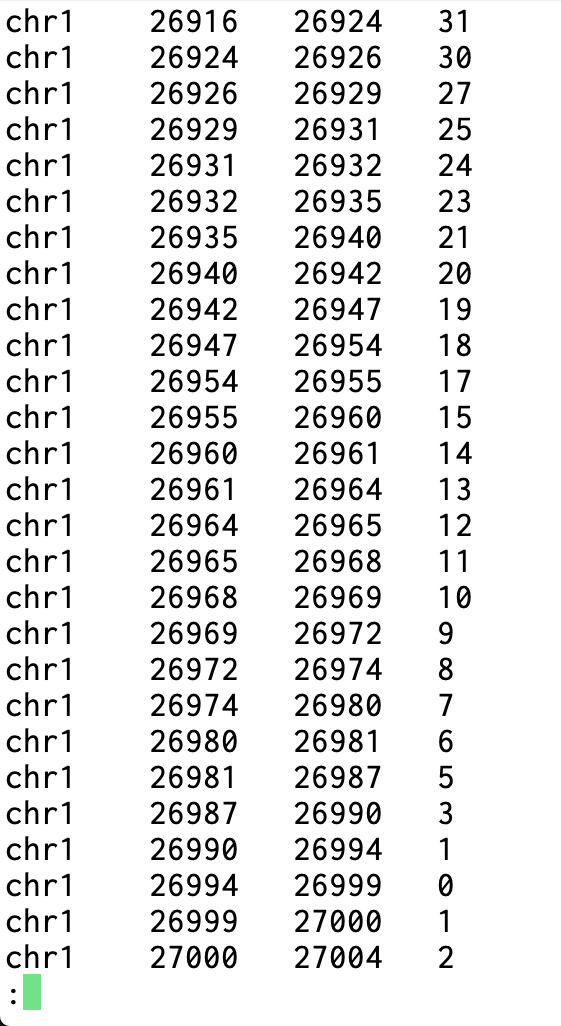
Mosdepth
Mosdepth는 전장 유전체 시퀀싱(WGS), 엑솜 시퀀싱, 표적 시퀀싱을 위한 고속 BAM/CRAM 커버리지 계산 도구입니다. Nim 언어로 개발되어 기존 도구들보다 월등히 빠른 성능을 제공하며, 메모리 효율적인 알고리즘을 통해 대용량 시퀀싱 데이터의 커버리지 분석을 신속하게 수행합니다. 이 도구는 독특한 염색체별 배열 기반 알고리즘을 사용합니다. 각 염색체에 대해 배열을 생성하고, 리드의 시작 위치에서는 값을 증가시키고 끝 위치에서는 감소시킨 후, 누적합을 계산하여 각 위치의 커버리지를 구합니다. 이 방법은 CIGAR 연산을 정확히 추적하여 리드의 정렬된 모든 부분을 포함하며, 겹치는 mate-pair를 이중 계산하지 않는 정교한 커버리지 측정을 제공합니다. Mosdepth는 단순한 커버리지 계산을 넘어서 다양한 고급 기능을 제공합니다. 윈도우 기반 분석, BED 파일 기반 영역별 분석, 임계값 기반 커버리지 통계, 그리고 quantize 기능을 통한 커버리지 구간 분할 등이 가능합니다. 특히 분포 계산은 런타임 증가 없이 자동으로 수행되며, 다중 스레드를 활용한 압축 해제 최적화로 대규모 데이터셋에서도 뛰어난 성능을 발휘합니다.
주요사항
- > 커버리지 품질 기준 - 평균 깊이 : WGS 30X 이상 권장 - 균일성 : CV(변동계수) < 0.3 - 10X 이상 비율 : >90% - 30X 이상 비율 : >80%
실행 명령어 예시
$PROGRAM_DIR/mosdepth –t 6 $OUTPUT_PREFIX $INPUT_BAM
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | BAM 파일이 있는 디렉토리 경로 | |
| Output | Folder | output_dir | path/to/output_dir | 실행 후 결과물을 저장할 디렉토리 경로 |
결과
Conpair
Conpair는 인간 종양-정상(Tumor-Normal) 쌍 연구를 위해 특별히 설계된 빠르고 견고한 방법론으로, concordance 검증(동일 개체 유래 확인)과 교차 개체 오염 수준 추정을 전장 유전체 및 엑솜 시퀀싱 실험에서 수행합니다. 이 도구의 가장 중요한 특징은 종양 샘플의 오염 추정 방법이 복제수 변화(copy number changes)에 영향받지 않으며, 0.1%라는 매우 낮은 오염 수준까지 검출할 수 있다는 점입니다. Conpair는 New York Genome Center에서 개발된 도구로, 미리 선택된 유전체 위치의 집합을 기반으로 작동합니다. 이 마커들은 1000 Genomes Project Phase 3 데이터에서 선별된 것으로, MAF(Minor Allele Frequency) ≥ 0.4, LD(Linkage Disequilibrium) ≤ 0.8 기준을 충족하는 고품질 SNV들입니다. 두 단계 프로세스를 통해 작동하는데, 먼저 GATK를 사용하여 각 샘플에 대해 미리 정의된 마커 위치에서 pileup을 생성하고, 그 다음 이 pileup 데이터를 분석하여 concordance와 contamination을 계산합니다. 특히 임상 응용에서 중요한 점은 종양 샘플에서 0.5%라는 매우 낮은 오염 수준도 체세포 변이 호출(somatic mutation calling)에 심각한 영향을 미쳐 특이도를 크게 감소시킨다는 것입니다. 반면 정상 샘플의 교차 개체 오염은 체세포 변이 호출에 상대적으로 경미한 영향을 미칩니다.
주요사항
- 1. Concordance(일치성) 평가 기준 > 정상 범위: - 99-100%: 동일 개체에서 유래한 concordant 샘플 - 복제수 변화 영향 제거: --normal_homozygous_markers_only 옵션 사용 권장 > 주의 범위: - 80-99%: 오염 및/또는 복제수 변화의 영향 (정상 동형접합 마커만 사용하지 않은 경우) - 검토 필요: 샘플 품질 및 분석 파라미터 재확인 요구 > 문제 범위: - ~40%: 서로 다른 개체에서 유래한 discordant 샘플 - 샘플 스왑: 라벨링 오류 또는 교차 오염 의심
- 2. Contamination(오염도) 평가 기준 > 종양 샘플 오염: - < 0.5%: 허용 가능한 수준 - ≥ 0.5%: 체세포 변이 호출에 심각한 영향, 특이도 현저히 감소 - ≥ 2.0%: 분석 결과 신뢰성 크게 손상 - ≥ 5.0%: 분석 재수행 권장 > 정상 샘플 오염: - < 2.0%: 체세포 변이 호출에 경미한 영향 - ≥ 2.0%: 위양성 증가 가능성 - ≥ 5.0%: 주의 깊은 해석 필요
- 3. 임상적 의미 > 체세포 변이 호출에 미치는 영향: - 종양 오염: 변이 검출 민감도 감소, 위음성 증가 - 정상 오염: 위양성 증가, 특이도 감소 - 0.1% 검출 한계: 매우 낮은 오염도도 정확한 정량 가능 > 권장 조치: - 재수행 기준: 종양 오염 ≥ 2%, 정상 오염 ≥ 5% - 필터링 강화: 중간 수준 오염시 변이 호출 임계값 상향 조정 - 원인 분석: 샘플 수집, 처리, 라이브러리 제작 과정 검토
실행 명령어 예시
1. GATK Pileup Generation: Generate pileups at predefined marker positions for each # Generate tumor sample pileup ${CONPAIR_DIR}/scripts/run_gatk_pileup_for_sample.py -B TUMOR.bam -O TUMOR_pileup # Generate normal sample pileup ${CONPAIR_DIR}/scripts/run_gatk_pileup_for_sample.py -B NORMAL.bam –O NORMAL_pileup 2. Concordance Validation: Verify that both samples are derived from the same individual ${CONPAIR_DIR}/scripts/verify_concordance.py -T TUMOR_pileup –N NORMAL_pileup 3. Contamination Estimation: Estimate cross-contamination levels between tumor and normal samples ${CONPAIR_DIR}/scripts/estimate_tumor_normal_contamination.py -T TUMOR_pileup -N NORMAL_pileup
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | BAM 파일이 있는 디렉토리 경로 | |
| Output | Folder | output_dir | path/to/output_dir | 실행 후 결과물을 저장할 디렉토리 경로 | |
| Option | File | ref_genome | hg38 | Reference sequence FASTA 파일 경로 |
결과
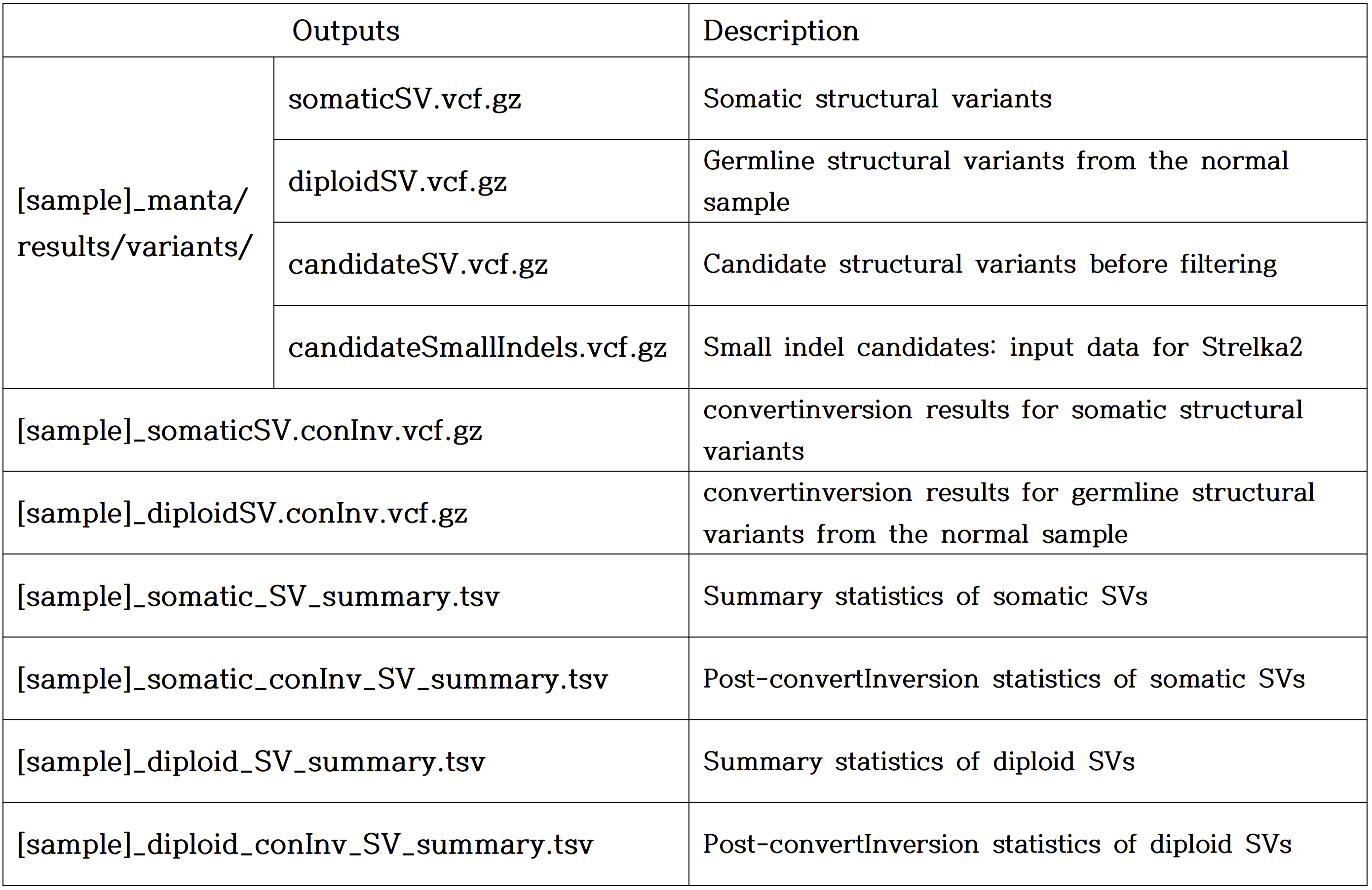
Manta Tumor-Normal
Manta는 Illumina에서 개발한 종양-정상 쌍(Tumor-Normal pair) 시퀀싱 데이터 전용 구조 변이(Structural Variants, SVs) 검출 도구로, 체세포 구조 변이를 정확하고 민감하게 찾아내는 데 특화되어 있습니다. 이 도구는 split-read 분석, discordant read-pair 분석, 그리고 local assembly를 결합한 정교한 알고리즘을 사용하여 결손(deletion), 삽입(insertion), 역위(inversion), 전좌(translocation), 중복(duplication) 등 다양한 종류의 대규모 유전체 구조 변화를 검출합니다. Manta의 Tumor-Normal 모드는 종양 샘플과 정상 샘플을 동시에 분석하여 체세포 특이적인 구조 변이만을 선별적으로 검출합니다. 정상 샘플에서는 생식세포 구조 변이(germline SVs)를 식별하고, 종양 샘플에서는 체세포 구조 변이(somatic SVs)를 검출하여 이들을 구분합니다. 이 과정에서 높은 민감도와 특이도를 유지하면서도 위양성을 효과적으로 제거합니다. 도구는 multi-threading을 지원하여 대용량 전장 유전체 시퀀싱 데이터도 효율적으로 처리할 수 있으며, BAM과 CRAM 파일 형식을 모두 지원합니다. Paired-end와 single-end 시퀀싱 데이터를 처리할 수 있고, 다중 샘플 분석도 가능합니다. 특히 암 연구에서 중요한 종양 특이적 구조 변이를 정확하게 식별하여 암의 진행, 전이, 치료 저항성과 관련된 유전체 불안정성을 분석하는 데 필수적인 도구입니다.
주요사항
- > 구조 변이 유형별 특징 1. DEL (Deletion) - 결손 - 크기 범위: 50bp - 수 Mb - 검출 방법: Split-read + Discordant pair - 임상 의미: 유전자 전체 또는 일부 소실 - 예상 빈도: 개체당 약 4,000-6,000개 2. DUP (Duplication) - 중복 - 유형: Tandem duplication, Interspersed duplication - 크기 범위: 50bp - 수 Mb - 검출 방법: Read depth + Discordant pair - 임상 의미: 유전자 복제수 증가 - 예상 빈도: 개체당 약 500-1,000개 3. INV (Inversion) - 역위 - 유형: Paracentric, Pericentric - 크기 범위: 1kb - 수 Mb - 검출 방법: Split-read cluster 분석 - 임상 의미: 유전자 발현 또는 기능 변화 - 예상 빈도: 개체당 약 100-300개 4. TRA (Translocation) - 전좌 - 유형: Balanced, Unbalanced - 검출 방법: Discordant read-pair 분석 - 임상 의미: 염색체간 재배열 - 예상 빈도: 개체당 약 50-200개 5. INS (Insertion) - 삽입 - 유형: Mobile element, Novel sequence - 크기 범위: 50bp - 수 kb - 검출 방법: Split-read + Assembly - 임상 의미: 새로운 서열 삽입 - 예상 빈도: 개체당 약 500-1,500개
- > 품질 필터링 기준 1. PASS 필터 조건 - 최소 지지 리드: Split-read ≥ 3, Paired-read ≥ 3 - Genotype Quality: GQ ≥ 15 - Mapping Quality: 매핑된 리드의 평균 MAPQ ≥ 20 - Repeat Content: 반복 서열 영역 회피 - Assembly Quality: Local assembly 성공 2. 필터 태그 의미 - PASS: 모든 품질 기준 통과 - LowGQ: 낮은 유전형 품질 (GQ < 15) - LowGQX: 확장 품질 점수 부족 - HomRef: 참조 동형접합 (변이 없음) - NotGenotyped: 유전형 결정 실패
- > Manta 결과의 복잡한 역위(inversion) 변이의 convertInversion.py 처리를 통한 표준화된 형태로의 변환 전후 비교 예시 - 변환 전 (복잡한 형태): chr3 100000 . N ]chr3:200000]N . PASS SVTYPE=BND;MATEID=chr3_100000_2 chr3 200000 . N N[chr3:100000[ . PASS SVTYPE=BND;MATEID=chr3_100000_1 - 변환 후 (표준화): chr3 100000 . N <INV> . PASS SVTYPE=INV;SVLEN=100000;END=200000 - 원본 vs 변환후 구조 변이 수 비교 Original_INV: 8 complex_breakpoints Converted_INV: 12 standard_inversions 전체적으로 SV 4개 증가: 복잡한 BND가 단순한 INV로 병합됨
실행 명령어 예시
#1. Workflow Setup: Generate configuration files and working scripts for tumor-normal paired analysis python $PROGRAM_DIR/bin/configManta.py \ --normalBam $NORMAL_BAM \ --tumorBam $TUMOR_BAM \ --referenceFasta $REFERENCE_GENOME \ --runDir $SAMPLE_output_dir #2. Workflow Execution: Execute the configured workflow to perform actual SV detection python $SAMPLE_output_dir/runWorkflow.py -m local –j 6 #3. Convert INV represented by two breakends into a single INV record to enhance interpretability python $PROGRAM_DIR/libexec/convertInversion.py \ $PROGRAM_SAMTOOLS \ $REFERENCE_GENOME \ $RESULT_DIR/diploidSV.vcf.gz
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | BAM 파일이 있는 디렉토리 경로 | |
| Output | Folder | output_dir | path/to/output_dir | 실행 후 결과물을 저장할 디렉토리 경로 | |
| Option | Folder | ref_genome | hg38 | Reference sequence FASTA 파일 경로 |
결과
Strelka2
Strelka2는 Illumina에서 개발한 체세포 변이 검출 솔루션입니다. 이 도구는 암 유전체학 연구와 임상 진단 분야에서 핵심적인 역할을 담당하며, 종양과 정상 조직의 쌍 시퀀싱 데이터를 정교하게 비교 분석하여 암 특이적인 유전적 변화를 발굴하는 데 특화되어 있습니다. 특히 Strelka2는 단일 염기 변이(Single Nucleotide Variants, SNVs)와 작은 규모의 삽입/결손 변이(Insertions and Deletions, Indels)를 동시에 검출할 수 있는 통합 플랫폼으로 설계되었습니다. 이러한 변이들은 암의 발생, 진행, 전이 과정에서 핵심적인 역할을 하는 드라이버 변이(driver mutations)부터 치료 저항성과 관련된 변이까지 포괄하여, 개인 맞춤형 암 치료 전략 수립에 필수적인 정보를 제공합니다. 도구의 가장 큰 강점은 높은 민감도와 정확도를 동시에 확보한 베이지안 확률론적 접근법을 채택했다는 점입니다. 기존의 단순한 통계적 검정 방식을 넘어서, 시퀀싱 오류, PCR 증폭 오류, 정렬 아티팩트 등 다양한 기술적 노이즈를 지능적으로 구별하고 필터링하여 진정한 체세포 변이만을 선별해낼 수 있습니다. 또한 매우 낮은 변이 대립유전자 빈도(Variant Allele Frequency, VAF)를 가진 변이들도 신뢰성 있게 검출할 수 있어, 종양 내 이질성(intratumor heterogeneity)이나 순환 종양 DNA(circulating tumor DNA) 분석과 같은 고도의 정밀도가 요구되는 연구 영역에서도 활용 가능합니다.
주요사항
- 1. 기본 품질 기준 > PASS 필터 기준: - 모든 필터 통과: 가장 높은 신뢰도의 체세포 변이 - 베이지안 확률: 체세포 변이일 확률 ≥ 99.9% - 양방향 증거: 정상/종양 양쪽에서 일관된 증거 확인 > VAF (Variant Allele Frequency) 기준: - 체세포 SNV: 종양 ≥3%, 정상 <1% - 체세포 Indel: 종양 ≥5%, 정상 <2% - 최소 검출 한계: 1-2% VAF (충분한 깊이 조건하에) > 깊이(Coverage) 권장사항: - WGS 표준: 종양/정상 각각 30X 이상 - 고정밀 분석: 종양/정상 각각 60X 이상 - 최소 요구사항: 변이 위치에서 종양 20X, 정상 15X
- 2. Strelka2 특화 필터 시스템 > SNV 전용 필터: - QSS_ref: SNV 품질 점수 부족 (QSS < 임계값) - LowEVS: 변이 증거 점수 낮음 (EVS < 2.0) - BCNoise: 염기 조성 노이즈 (주변 서열 반복) - SpanDel: 삭제와 겹치는 영역의 SNV - SnpGap: 다른 변이와 너무 가까운 거리 (< 10bp) > Indel 전용 필터: - QSI_ref: Indel 품질 점수 부족 (QSI < 임계값) - LowDepth: 깊이 부족 (총 깊이 < 3) - RepeatRegion: 반복 서열 영역 (STR, homopolymer) - iHpol: Homopolymer run 관련 아티팩트 > 공통 필터: - MaxDepth: 과도한 깊이 (> 1000X, 반복 서열 의심) - NoPassedVariantGTs: 유효한 유전형 없음 - IndelConflict: 다른 indel과 충돌
실행 명령어 예시
# 1. Workflow Configuration python $PROGRAM_DIR/configureStrelkaSomaticWorkflow.py \ --normalBam $NORMAL_BAM \ --tumorBam $TUMOR_BAM \ --referenceFasta $REFERENCE_GENOME \ --runDir $output_dir # 2. Workflow Execution python $output_dir/runWorkflow.py -m local -j 6
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | 종양 및 정상 BAM 파일이 있는 디렉토리 경로 | |
| Input | Folder | indel_vcf_dir | path/to/indel_vcf_dir | manta의 candidate small indel VCF 파일의 디렉토리 | |
| Output | Folder | output_dir | path/to/output_dir | 실행 후 결과물을 저장할 디렉토리 경로 | |
| Option | File | ref_genome | hg38 | 참조 유전체 FASTA 파일 경로 | |
| Option | File | call_region_bed | hg38 | 분석 대상 영역 지정 BED |
결과
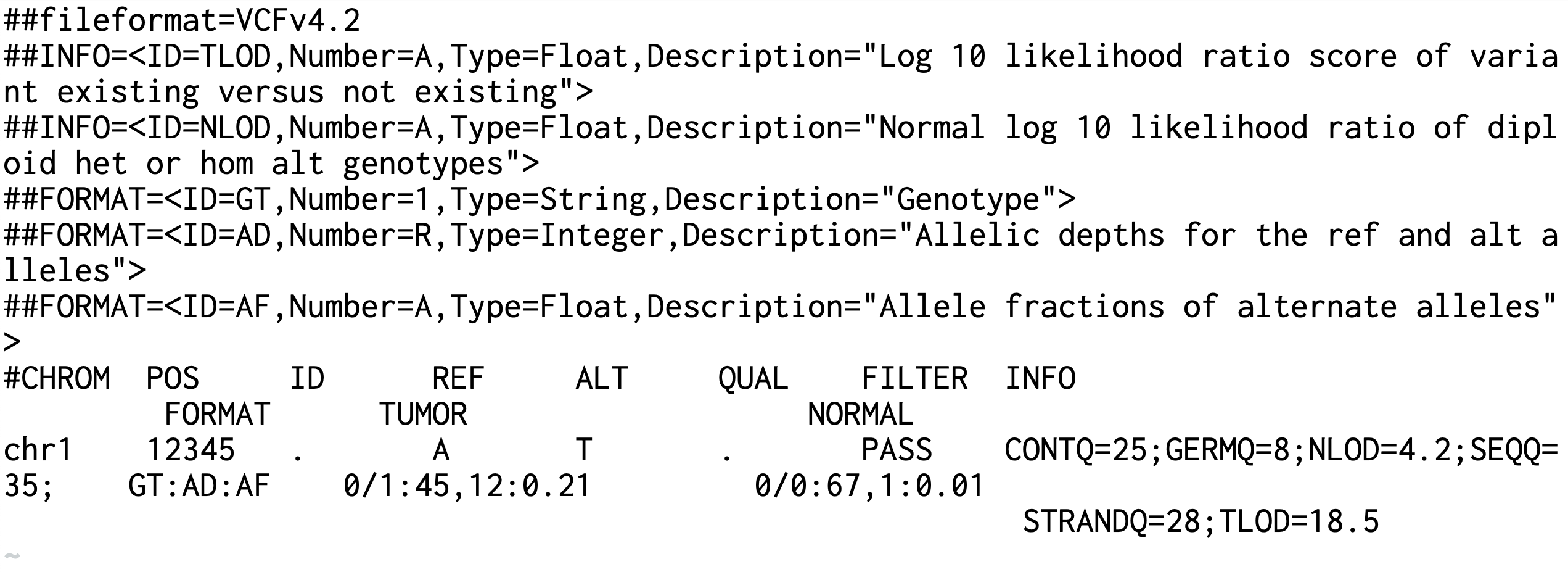
GATK Mutect2
GATK Mutect2는 Broad Institute에서 개발한 체세포 변이 검출 플랫폼으로, 암 유전체학 연구와 정밀 의학 분야에서 금본위제(gold standard)로 인정받고 있는 핵심 도구입니다. 이 도구는 GATK Best Practices 워크플로우의 핵심 구성 요소로서, 종양과 정상 조직 간의 미세한 유전적 차이를 베이지안 통계학적 프레임워크를 통해 정교하게 분석하여 진정한 체세포 변이만을 선별적으로 검출하는 데 특화되어 있습니다. 특히 Mutect2의 가장 큰 강점은 매우 엄격한 통계적 검정과 다층적 품질 관리 시스템을 통해 위양성(false positive) 결과를 최소화하면서도 임상적으로 중요한 저빈도 변이들을 놓치지 않는 균형잡힌 성능을 제공한다는 점입니다. 이 도구는 단일 염기 변이(SNVs)와 짧은 삽입/결손 변이(indels)를 동시에 검출할 수 있으며, 종양-정상 쌍(tumor-normal paired) 분석 모드뿐만 아니라 종양 단독(tumor-only) 분석 모드도 지원하여 다양한 연구 환경과 임상 상황에 유연하게 적용할 수 있습니다. Mutect2의 핵심 혁신은 정교한 베이지안 체세포 유전형 모델(Bayesian somatic genotyping model)과 함께 Panel of Normals(PoN) 기반의 체계적 아티팩트 제거, 실시간 오염도 추정(contamination estimation), 그리고 방향성 편향 보정(orientation bias correction) 등의 고급 기능들을 통합한 포괄적 분석 파이프라인을 제공한다는 것입니다. 이러한 다면적 접근법을 통해 시퀀싱 과정에서 발생할 수 있는 다양한 기술적 노이즈와 생물학적 혼재 요인들을 지능적으로 구별하고 필터링하여, 암의 진단, 예후 예측, 치료 반응 모니터링, 그리고 신약 개발 연구에 필수적인 고품질의 체세포 변이 정보를 제공합니다.
주요사항
- VCF INFO 컬럼에서 확인 가능한 품질 메트릭들의 해석 구간: > TLOD (Tumor Log-Odds): 변이 증거의 로그 우도비 - ≥20: 매우 강한 체세포 변이 증거 (고신뢰도) - 10-20: 강한 체세포 변이 증거 (중고신뢰도) - 6.3-10: 보통 체세포 변이 증거 (기본 통과 임계값) - 3-6.3: 약한 체세포 변이 증거 (추가 검증 필요) - <3: 매우 약한 증거 (일반적으로 필터링)
- > NLOD (Normal Log-Odds): 정상 시료에서의 변이 증거 - ≥5: 정상 시료에서 강한 변이 신호 (생식세포 변이 의심) - 2-5: 정상 시료에서 중간 변이 신호 (오염 또는 생식세포 변이) - 0-2: 정상 시료에서 약한 변이 신호 (낮은 VAF 체세포 변이 가능) - <0: 정상 시료에서 변이 증거 없음 (체세포 변이 지지)
- > GERMQ: 생식세포 변이 확률 (Phred scale) - ≥30: 생식세포 변이일 확률 99.9% (거의 확실한 생식세포 변이) - 20-30: 생식세포 변이일 확률 99-99.9% (높은 확률의 생식세포 변이) - 10-20: 생식세포 변이일 확률 90-99% (중간 확률의 생식세포 변이) - 3-10: 생식세포 변이일 확률 50-90% (낮은 확률, 추가 분석 필요) - <3: 생식세포 변이일 확률 <50% (체세포 변이 가능성 높음)
- > CONTQ: 오염도 보정 품질 점수 - ≥30: 오염도 보정이 매우 정확함 (높은 신뢰도) - 20-30: 오염도 보정이 정확함 (중간 신뢰도) - 10-20: 오염도 보정이 어느 정도 정확함 (주의 필요) - 3-10: 오염도 보정 불확실함 (낮은 신뢰도) - <3: 오염도 보정 매우 불확실함 (필터링 고려)
- > SEQQ: 시퀀싱 아티팩트 품질 점수 - ≥30: 시퀀싱 아티팩트 아닐 확률 99.9% (매우 높은 신뢰도) - 20-30: 시퀀싱 아티팩트 아닐 확률 99-99.9% (높은 신뢰도) - 10-20: 시퀀싱 아티팩트 아닐 확률 90-99% (중간 신뢰도) - 3-10: 시퀀싱 아티팩트 아닐 확률 50-90% (낮은 신뢰도) - <3: 시퀀싱 아티팩트일 가능성 높음 (필터링 권장)
- > STRANDQ: 스트랜드 편향 품질 점수 - ≥30: 스트랜드 편향 없음 (매우 균등한 분포) - 20-30: 경미한 스트랜드 편향 (허용 가능한 수준) - 10-20: 중간 스트랜드 편향 (주의 깊은 검토 필요) - 3-10: 강한 스트랜드 편향 (아티팩트 가능성 높음) - <3: 매우 강한 스트랜드 편향 (필터링 권장)
실행 명령어 예시
# Group 1: Large Chromosomes -L chr1 -L chr2 -L chr3 -L chr4 -L chr5 # Group 2: Medium Chromosomes -L chr6 -L chr7 -L chr8 -L chr9 -L chr10 -L chr11 -L chr12 # Group 3: Small Chromosomes and Sex Chromosomes -L chr13 -L chr14 -L chr15 -L chr16 -L chr17 -L chr18 -L chr19 -L chr20 -L chr21 -L chr22 -L chrX -L chrY –L chrM # Concurrent Execution of 3 Groups java -Xms7g -Xmx7g -jar gatk.jar Mutect2 -I normal.bam -I tumor.bam -O group1.vcf -R ref.fa -L chr1-5 & java -Xms7g -Xmx7g -jar gatk.jar Mutect2 -I normal.bam -I tumor.bam -O group2.vcf -R ref.fa -L chr6-12 & java -Xms7g -Xmx7g -jar gatk.jar Mutect2 -I normal.bam -I tumor.bam -O group3.vcf -R ref.fa -L chr13-M & wait
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | BAM 파일이 있는 디렉토리 경로 | |
| Output | Folder | output_dir | path/to/output_dir | 실행 후 결과물을 저장할 디렉토리 경로 | |
| Option | File | ref_genome | hg38 | 참조 유전체 FASTA 파일 경로 | |
| Option | File | germline_resource | somatic-hg38_af-only-gnomad.hg38.vcf.gz | 인구 집단 내에서 흔히 관찰되는 정상적인 생식세포(germline) 변이(SNP, Indel) 데이터베이스를 제공 |
결과
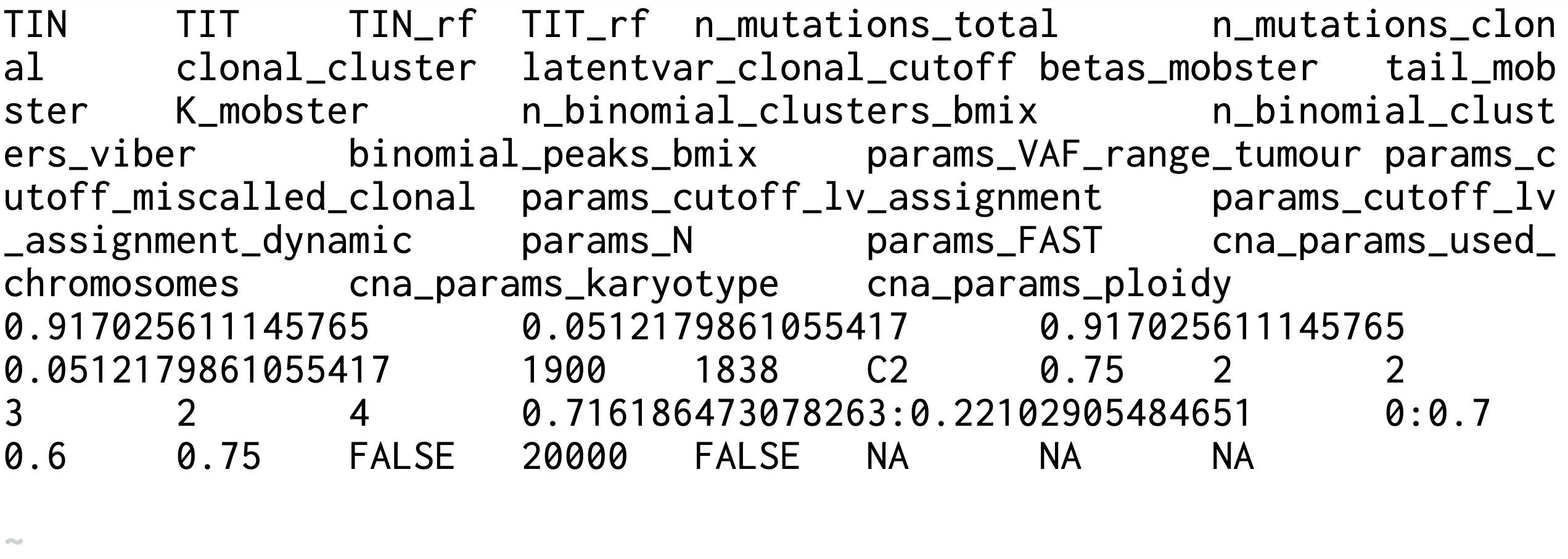
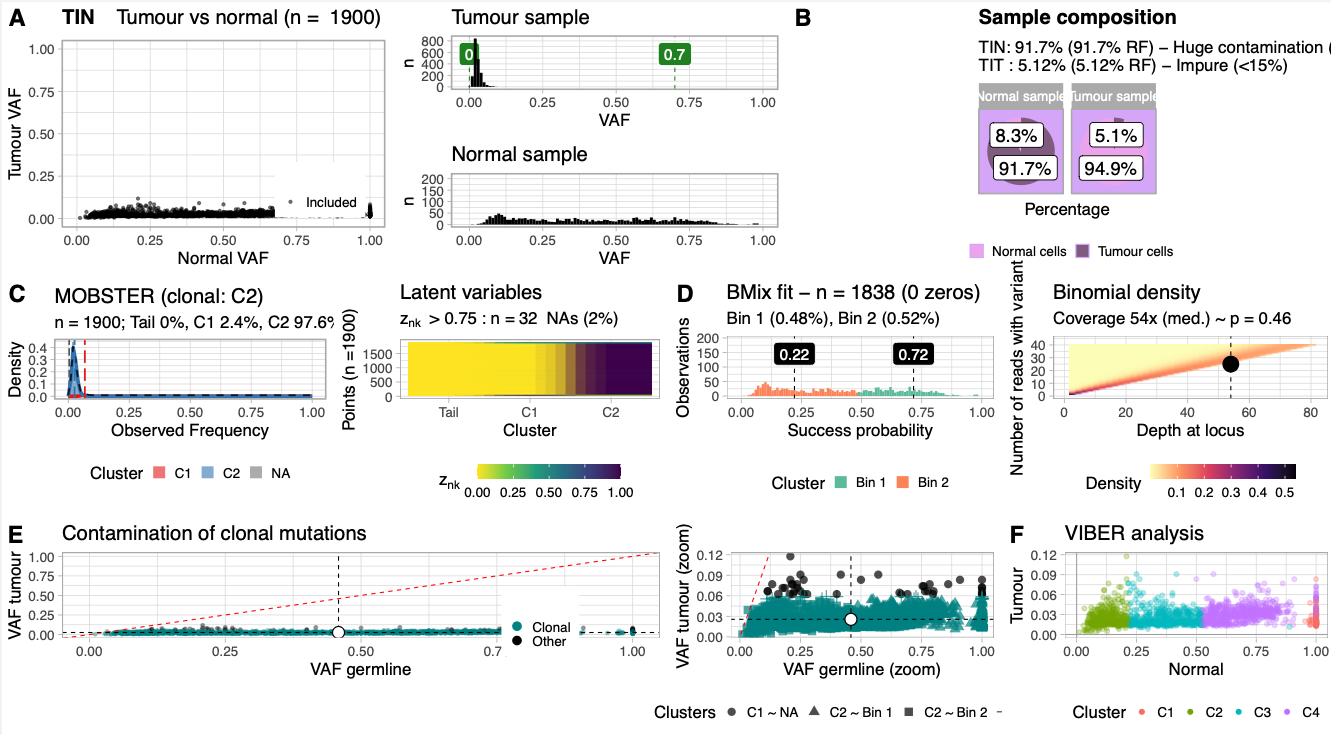
TINC
TINC(Tumor-in-Normal Contamination)는 Giulio Caravagna 연구팀에서 개발한 첨단 종양 오염도 분석 플랫폼으로, 암 유전체학 연구에서 가장 까다로운 문제 중 하나인 교차 샘플 오염(cross-sample contamination)을 정밀하게 정량화하는 혁신적인 도구입니다. 이 툴은 매칭된 종양-정상 쌍(tumor-normal paired) 시료에서 TIN(Tumor-in-Normal)과 TIT(Tumor-in-Tumour) 점수를 동시에 추정하여, 샘플 채취, 처리, 시퀀싱 과정에서 발생할 수 있는 다양한 형태의 오염을 체계적으로 감지하고 정량화합니다. 특히 TINC의 핵심 강점은 단순한 오염도 측정을 넘어서, 복잡한 베이지안 통계학적 모델링을 통해 종양의 클론 구조(clonal architecture)와 서브클론 진화(subclonal evolution) 패턴을 동시에 분석한다는 점입니다. 이 도구는 MOBSTER(변이 대립유전자 빈도 클러스터링), BMix(정상 시료 베이지안 혼합 모델), VIBER(변분 베이지안 추론) 등의 고급 알고리즘을 통합적으로 활용하여, 전통적인 방법으로는 구별하기 어려운 미세한 오염 신호까지도 신뢰성 있게 검출할 수 있습니다. 또한 복제수 변이(CNA) 정보를 선택적으로 활용하여 분석의 정확도를 한층 더 향상시킬 수 있습니다. TINC는 특히 액체 생검(liquid biopsy), 최소 잔존 질환(MRD) 모니터링, 종양 이질성 연구, 그리고 임상 시료의 품질 관리에서 필수적인 역할을 담당합니다. 종양 순도가 낮은 시료나 정상 조직에 미세한 종양 세포 침윤이 의심되는 경우, TINC의 정밀한 오염도 분석 결과는 후속 체세포 변이 분석의 신뢰성을 크게 향상시키고, 위양성 및 위음성 결과를 최소화하여 개인 맞춤형 치료 전략 수립에 핵심적인 정보를 제공합니다.
주요사항
- > Mutect2 또는 Strelka2에서 생성된 Tumor-Normal pair의 분석 결과로서, 체세포 변이 VCF 파일 - Bio-Express 파이프라인에서는 파일명에 “somatic”이라는 키워드가 포함되어 있으면 인식하여 분석 시작 - 최소 50개 이상의 변이 필요. 100개 이상의 PASS 변이 권장.
입력 데이터 예제
실행 명령어 예시
print(sampled_data) load_TINC_input(x = sampled_data$data, cna = sampled_data$cna) TINC_fit = autofit(sampled_data$data, cna = NULL, FAST = TRUE) plot(TINC_fit) plot_full_page_report(TINC_fit)
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | 체세포 변이 분석 결과 VCF 파일이 있는 디렉토리 경로(파일명에 “somatic“ 키워드 포함 필요) | |
| Output | Folder | output_dir | path/to/output_dir | 실행 후 결과물을 저장할 디렉토리 경로 |
결과
SAMtools_flagstat
SAMtools flagstat는 BAM, SAM, CRAM 파일의 FLAG 정보를 기반으로 정렬 통계를 계산하고 요약 보고서를 제공하는 품질 관리 도구입니다. 이 도구는 입력 파일을 한 번 완전히 통과하면서 FLAG 필드의 비트 플래그를 기준으로 13개 카테고리의 통계를 산출하여 표준 출력으로 결과를 제공합니다. FLAG 정보는 SAM 형식 사양서에 정의된 비트 플래그로, 각 리드의 정렬 상태와 특성을 나타내는 중요한 메타데이터입니다. flagstat는 전체 서열 수, 매핑된 서열 수, 쌍으로 매핑된(properly paired) 서열 수, 중복(duplicates) 리드 수, 주 정렬(primary), 보조 정렬(secondary), 보완 정렬(supplementary) 등의 세부 통계를 제공합니다. 각 카테고리는 QC 통과(QC-passed)와 QC 실패(QC-failed) 리드로 구분되어 "#PASS + #FAIL" 형태로 출력되며, 매핑 비율, 적절한 페어링 비율 등의 백분율 정보도 함께 제공됩니다. 또한 기본 형식 외에도 TSV(탭으로 구분된 값)와 JSON 형식으로 출력할 수 있어 스프레드시트 소프트웨어나 다른 분석 도구에서 쉽게 활용할 수 있습니다. 이러한 통계 정보는 시퀀싱 실험의 품질을 평가하고, 정렬 과정의 성공률을 확인하며, 데이터의 전반적인 특성을 파악하는 데 필수적인 지표로 활용됩니다. 특히 매핑률이 낮거나 적절하게 페어링되지 않은 리드의 비율이 높은 경우, 실험 조건이나 분석 파라미터를 재검토할 필요가 있음을 시사합니다. Bio-Express의 Whole Genome Sequencing Pipeline 분석과정에서 SAMtools flagstat는 입력(input) 데이터로 BAM 파일을 사용하며, 출력(output) 데이터로는 정렬 통계가 포함된 텍스트 형태의 요약 보고서를 생성합니다.
실행 명령어 예시
$PROGRAM_DIR/samtools flagstat -@ 6 \ -I $INPUT_BAM \ > $OUTPUT_FILE
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | path/to/input_dir | BAM 파일이 있는 디렉토리 경로 | |
| Output | Folder | output_dir | path/to/output_dir | 실행 후 결과물을 저장할 디렉토리 경로 |

.png)
.png)
.png)
.png)
.png)
.png)
.png)
.png)
.png)
.png)
.png)
.png)
.png)
.png)


.png)

.png)




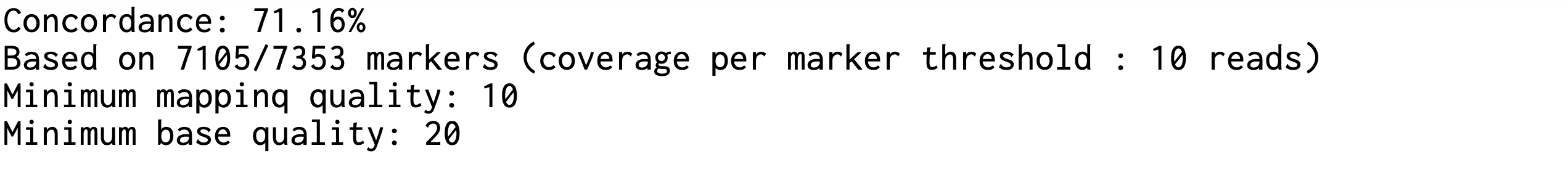

.png)
.png)
.png)
.png)